Сборка генома — процесс объединения большого количества коротких фрагментов ДНК (ридов) в одну или несколько длинных последовательностей (контигов и скаффолдов) в целях восстановления последовательностей ДНК хромосом, из которых возникли эти фрагменты в процессе секвенирования.
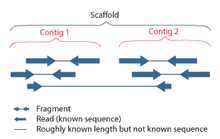
Сборка генома является очень сложной вычислительной задачей, в частности, осложнённой тем, что геномы часто содержат большое количество одинаковых повторяющихся последовательностей (так называемые геномные повторы). Эти повторы могут быть длиной в несколько тысяч нуклеотидов, а также встречаться в тысяче различных мест в геноме. Особенно богаты повторами большие геномы растений и животных, в том числе геном человека.
Алгоритмические подходы
правитьСуществует два подхода для сборки геномов — основанный на перекрытии overlap-layout-consensus (применяется для длинных фрагментов), а также основанный на графах де Брёйна (применяется для коротких фрагментов) [1][2].
Overlap-Layout-Consensus
правитьПри секвенировании методом дробовика все ДНК организма сначала разрезают на миллионы маленьких фрагментов до 1000 нуклеотидов в длину. Затем алгоритмы сборки генома рассматривают полученные фрагменты одновременно, находя их перекрытия (overlap), объединяя их по перекрытиям (layout) и исправляя ошибки в объединённой строке (consensus). Данные шаги могут повторяться несколько раз в процессе сборки.
Данный подход был наиболее распространён для сборки геномов до появления секвенирования следующего поколения.
Графы де Брёйна
правитьС развитием технологий секвенирования следующего поколения получение фрагментов стало на порядок дешевле, но размер фрагментов стал меньше (до 150 нуклеотидов), а количество ошибок при чтении фрагментов увеличилось (до 3 %). При сборке таких данных получили распространение методы[3], основанные на графах де Брёйна.
Доступные сборщики
правитьСписок популярных геномных сборщиков:
| Название | Поддерживаемые технологии | Авторы | Представлен | Обновлён | Лицензия* | Домашняя страница |
|---|---|---|---|---|---|---|
| ABySS | Solexa, SOLiD | Simpson, J. et al. | 2008 | 2011 | NC-A | ссылка |
| ALLPATHS-LG | Solexa, SOLiD | Gnerre, S. et al. | 2011 | 2011 | OS | ссылка |
| CLC Genomics Workbench | Sanger, 454, Solexa, SOLiD | CLC bio | 2008 | 2010 | C | ссылка |
| Euler | Sanger, 454 (,Solexa ?) | Pevzner, P. et al. | 2001 | 2006 | (C / NC-A?) | ссылка |
| Euler-sr | 454, Solexa | Chaisson, MJ. et al. | 2008 | 2008 | NC-A | ссылка |
| IDBA | Sanger,454,Solexa | Yu Peng, Henry C. M. Leung, Siu-Ming Yiu, Francis Y. L. Chin | 2010 | 2010 | (C / NC-A?) | ссылка |
| MIRA | Sanger, 454, Solexa | Chevreux, B. | 1998 | 2011 | OS | ссылка |
| Newbler | 454, Sanger | 454/Roche | 2009 | 2009 | C | ссылка |
| SOPRA | Illumina, SOLiD, Sanger, 454 | Dayarian, A. et al. | 2010 | 2011 | OS | ссылка |
| SOAPdenovo | Solexa | Li, R. et al. | 2009 | 2009 | OS | ссылка |
| SPAdes | Illumina, Solexa | Bankevich, A et al. | 2012 | 2012 | OS | ссылка |
| Velvet | Sanger, 454, Solexa, SOLiD | Zerbino, D. et al. | 2007 | 2009 | OS | ссылка |
| Canu | PacBio, Oxford Nanopore | Koren, S. et al. | 2017 | 2020 | OS | ссылка |
| *Licences: OS = Open Source; C = Коммерческая; C / NC-A = Коммерческая, но бесплатна для использования в некоммерческих и научных целях; Скобки = неизвестно, но скорее всего C / NC-A | ||||||
Примечания
править- ↑ Zhenyu Li et al. Comparison of the two major classes of assembly algorithms: overlap–layout–consensus and de-bruijn-graph (англ.) // Briefings in Functional Genomics : journal. — 2012. — Vol. 11, no. 1. — P. 25—37. — doi:10.1093/bfgp/elr035.
- ↑ Miller J. R., Koren S., Sutton G. Assembly algorithms for next-generation sequencing data (англ.) // Genomics : journal. — Academic Press, 2010. — Vol. 95, no. 6. — P. 315—327. Архивировано 22 января 2022 года.
- ↑ Pavel A. Pevzner, Haixu Tang, Michael S. Waterman. An Eulerian path approach to DNA fragment assembly (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2001. — Vol. 98, no. 17. — P. 9748—9753. — doi:10.1073/pnas.171285098. Архивировано 25 августа 2014 года.