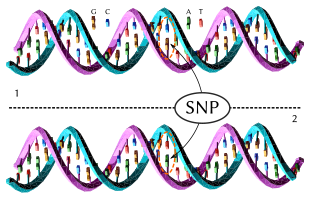
Однонуклеотидный полиморфизм (ОНП; англ. Single Nucleotide Polymorphism, SNP, произносится как снип, в современной иностранной литературе всё чаще заменяется на SNV - Single Nucleotide Variant) — отличия последовательности ДНК размером в один нуклеотид (A, T, G или C) в геноме (или в другой сравниваемой последовательности) представителей одного вида или между гомологичными участками гомологичных хромосом. Применяется в качестве генетических ма́ркеров для изучения неравновесного сцепления локусов и полногеномного поиска ассоциаций (GWAS).
Описание
правитьЕсли две последовательности ДНК — AAGCCTA и AAGCTTA — отличаются на один нуклеотид, в таком случае говорят о существовании двух аллелей: C и T. Однонуклеотидные полиморфизмы (SNPs) возникают в результате точечных мутаций.
Однонуклеотидный полиморфизм (наряду с полиморфизмом длин рестрикционных фрагментов (RFLP) и ПДАФ (AFLP)) широко используют в качестве молекулярно-генетических меток (маркеров), например, для построения кладограмм молекулярно-генетической систематики на основе дивергенции (расхождения) гомологичных участков ДНК в филогенезе. В данной области наиболее часто используются спейсеры генов рибосомальной РНК. Ввиду того, что мутации в данных спейсерах не сказываются на структуре конечных продуктов гена (теоретически они не влияют на жизнеспособность), в первом приближении постулируется прямая зависимость между степенью полиморфизма и филогенетическим расстоянием между организмами.
Номенклатура
правитьЕдиной номенклатуры для SNPs нет: часто существуют несколько различных вариантов названия для одного конкретно выбранного SNP, к какому-то согласию в этом вопросе прийти пока не удается. Один из подходов — писать SNPs с префиксом, точкой и знаком «больше, чем», показывающим нуклеотид или аминокислоту дикого типа и измененную (например, c.76A>T)[1].
Разнообразие SNPs
правитьОднонуклеотидный полиморфизм встречается в пределах кодирующих последовательностей генов, в некодирующих участках или в участках между генами. SNPs, встречающиеся в кодирующих участках, могут не менять аминокислотную последовательность белка из-за вырожденности генетического кода.
Однонуклеотидные полиморфизмы кодирующих участков бывают двух типов: синонимические и несинонимические. Синонимические SNPs оставляют аминокислотную последовательность белка без изменения, тогда как несинонимические SNPs изменяют её. Несинонимические SNPs можно разделить на замены типа missense и nonsense. Однонуклеотидный полиморфизм, встречающийся в некодирующих участках гена, возможно, влияет на генетический сплайсинг, деградацию мРНК, связывание транскрипционных факторов.
Примеры
править- rs6311 и rs6313 — SNP в гене HTR2A на 13-й хромосоме человека[2], для которых установлена связь с частотой суицидального и агрессивного поведения.
- rs3091244 — пример трёхаллельного SNP в гене CRP на 1-й хромосоме человека[3], варианты которого, предположительно, влияют на концентрацию C-реактивного белка в плазме крови.
- rs148649884 и rs138055828 в гене FCN1[англ.] кодируют M-фиколин[англ.], у которого отсутствует способность рекомбинантного M-фиколина к связыванию лиганда[4].
Области применения
правитьРазнообразием последовательностей ДНК у людей, возможно, объясняется то, как у них происходит течение различных заболеваний, реакции в ответ на патогены, прием лекарств, вакцин и т. п. Огромное значение SNPs в биомедицинских исследованиях состоит в том, что их используют для сравнения участков генома между исследуемыми группами (например, одна группа — люди с определенным заболеванием, а вторая — без него)[5].
Однонуклеотидные полиморфизмы также используются в GWAS — при генетическом картировании как маркеры с высоким разрешением, благодаря их количеству и стабильной наследуемости в ряду поколений. Знание об однонуклеотидном полиморфизме, вероятно, поможет в понимании фармакокинетики и фармакодинамики действия различных лекарств на человека. Широкий спектр заболеваний, такие как рак, инфекционные аутоиммунные заболевания, серповидноклеточная анемия и многие другие, возможно, возникают из-за однонуклеотидного полиморфизма[6].
Методы, основанные на выявлении однонуклеотидных полиморфизмов, получили также широкое распространение в других областях биологии и применительно к сельскохозяйственным видам[7].
Знание о наличие тех или иных SNPs также помогает учёным определять гаплотипы.
Базы данных
правитьДля SNPs существует большое количество баз данных. Ниже приведены некоторые из них.
- dbSNP[англ.][8] — база данных SNPs, свободный общественный архив, содержащий данные по наследственной изменчивости различных видов, разработанный и поддерживаемый NCBI (National Center for Biotechnology Information — Национальный центр биотехнологической информации США). Хотя такое название базы данных подразумевает, что там собран только один класс полиморфизмов, а именно SNPs, на самом деле она содержит большое количество информации и о других молекулярных изменениях в аминокислотных последовательностях. dbSNP была создана в сентябре 1998 года в дополнение к GenBank, в котором представлены нуклеотидные и аминокислотные последовательности, находящиеся в свободном доступе[9]. К 2010 году dbSNP содержала свыше 184 миллионов последовательностей, представляя более 64 миллионов различных вариантов для 55 организмов, включая Homo sapiens, Mus musculus, Oryza sativa и множество других[10].
- SNPedia[англ.] — биоинформатический вики-сайт, который служит как база данных SNPs. Каждая статья про отдельные SNPs предоставляет собой краткое описание, ссылки на научные статьи, и, кроме того, информацию о ДНК-микрочипе, содержащем однонуклеотидный полиморфизм данного типа. SNPedia помогает в интерпретации результатов собственной генетической информации с помощью таких программ как, например, Promethease, 23andMe, Navigenics[англ.], deCODEme[англ.] или Knome[англ.][11]. SNPedia была создана и поддерживается генетиком Грегом Ленноном и программистом Майком Кариазо. К 14 сентября 2017 года в базе данных содержалось 107 125 однонуклеотидных полиморфизмов[12].
- База данных GWAS Central[англ.] выдает краткое содержание объединенных данных в одном или нескольких полногеномных исследованиях. В этой базе данных представлено наиболее полное собрание p-value ассоциаций. GWAS Central использует мощные графические и текстовые методы представления данных для открытия и одновременной визуализации многих однонуклеотидных полиморфизмов. Исследователям также предоставляется возможность просматривать их персональные данные рядом с выбранными. Кроме того, данные находятся в свободном доступе для скачивания их научными сообществами.
- Международный проект HapMap — организация, целью которой является развитие карты гаплотипов человеческого генома, которая будет описывать общие паттерны генетической изменчивости у людей. HapMap — основной ресурс для выявления генетической изменчивости, влияющую на здоровье, факторы окружающей среды и т. п. Вся предоставляемая информация находится в свободном доступе. Этот проект — результат сотрудничества различных групп ученых из Канады, Китая, Японии, Нигерии, Великобритании и США, окончательная версия которого увидела свет весной 2009 года. В определенном участке генома располагается набор свободных SNPs (Tag SNPs[англ.]), которые хорошо коррелируют со всеми остальными SNPs в данном участке. Далее, изучив аллели свободных SNPs, можно с большей вероятностью определить гаплотип индивидуума. Так определяют гаплотипы у нескольких представителей (некоторые болеют определенным заболеванием, а другие нет), а потом, сравнивая две группы, определяют наиболее вероятное расположение SNPs и гаплотипов, которые вовлечены в заболевание.
- MirSNP — база данных однонуклеотидных полиморфизмов, изменяющих сайты связывания микроРНК. В ней содержится 12 846 SNPs, включая 1940 SNPs в пре-микроРНК[13].
Методы исследования SNPs
правитьАналитические методы открытия новых SNPs и обнаружения уже известных SNPs включают:
1. Гибридизационные методы
- Принцип молекулярных маяков[англ.].
- Суть этого принципа в том, что концы пробы (на которых находятся соответственно метка и тушитель флуоресценции) комплементарны друг другу. В результате при температуре отжига праймеров они схлопываются и образуют структуру типа «ручки сковородки» (шпильки — stem-loop), где зона комплементарности пробы с матрицей находится в петле. При гибридизации пробы с матрицей вторичная структура разрушается, флуоресцентная метка и тушитель расходятся в разные стороны, и флуоресценция от метки может быть детектирована.
- Детекция с помощью микрочипов.
- Динамическая аллель-специфическая гибридизация.
2. Ферментативные методы
- Полиморфизм длин рестрикционных фрагментов.
- Методы, основанные на ПЦР.
- Удлинение праймеров.
- TaqMan[англ.]-пробы.
- Лигирование[англ.] олигонуклеотидов.
- Капиллярный электрофорез[14].
3. Методы, основанные на физических свойствах ДНК:
- Одноцепочечный конформационный полиморфизм[англ.] (SSCP).
- Электрофорез по градиенту температур[англ.] (TGGE).
- Высокоэффективная жидкостная хроматография в денатурирующих условиях.
- Масс-спектрометрия[15].
4. Секвенирование ДНК[16]. Для картирования SNPs на протяжении всего генома сейчас применяют методы секвенирования нового поколения и дальнейшую биоинформатическую обработку данных, полученных в ходе секвенирования.
См. также
правитьПримечания
править- ↑ Den Dunnen J. T. Recommendations for the description of sequence variants (англ.) // Human Genome Variation Society : journal. — 2008.
- ↑ Giegling I., Hartmann A. M., Möller H. J., Rujescu D. Anger- and aggression-related traits are associated with polymorphisms in the 5-HT-2A gene (англ.) // Journal of Affective Disorders[англ.] : journal. — 2006. — November (vol. 96, no. 1—2). — P. 75—81. — doi:10.1016/j.jad.2006.05.016. — PMID 16814396.
- ↑ Morita, Akihiko; Nakayama, Tomohiro; Doba, Nobutaka; Hinohara, Shigeaki; Mizutani, Tomohiko; Soma, Masayoshi. Genotyping of triallelic SNPs using TaqMan PCR (англ.) // Molecular and Cellular Probes[англ.] : journal. — 2007. — Vol. 21, no. 3. — P. 171—176. — doi:10.1016/j.mcp.2006.10.005. — PMID 17161935.
- ↑ Ammitzbøll, Christian Gytz; Kjær, Troels Rønn; Steffensen, Rudi; Stengaard-Pedersen, Kristian; Nielsen, Hans Jørgen; Thiel, Steffen; Bøgsted, Martin; Jensenius, Jens Christian. Non-Synonymous Polymorphisms in the FCN1 Gene Determine Ligand-Binding Ability and Serum Levels of M-Ficolin (англ.) // PLoS ONE : journal. — 2012. — 28 November (vol. 7, no. 11). — P. e50585. — doi:10.1371/journal.pone.0050585. Архивировано 25 сентября 2015 года.
- ↑ Carlson, Bruce (15 June 2008). "SNPs — A Shortcut to Personalized Medicine". Genetic Engineering & Biotechnology News. 28 (12). Mary Ann Liebert, Inc. Архивировано 26 декабря 2010. Дата обращения: 6 июля 2008.
(subtitle) Medical applications are where the market's growth is expected
- ↑ INGRAM VM. A specific chemical difference between the globins of normal human and sickle-cell anaemia haemoglobin. (англ.) // Nature. — 1956. — 13 October (vol. 178, no. 4537). — P. 792—794. — doi:10.1038/178792a0. — PMID 13369537. Архивировано 27 февраля 2020 года.
- ↑ Romanov M. N., Miao Y., Wilson P. W., Morris A., Sharp P. J., Dunn I. C. (1999-05-16). "Detection and assay of polymorphism in reproductive gene loci in a commercial broiler breeder population for use in association studies". Proceedings. Conference «From Jay Lush to Genomics: Visions for Animal Breeding and Genetics» (Ames, 16—18 May 1999). Ames, IA, USA: Iowa State University. p. 155. OCLC 899128332. Abstract 15. Архивировано из оригинала 14 марта 2005. Дата обращения: 14 марта 2005.
{{cite conference}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) (англ.) - ↑ Wheeler D. L., Barrett T., Benson D. A., Bryant S. H., Canese K., Chetvernin V., Church D. M., DiCuccio M., Edgar R., Federhen S., Geer L. Y., Kapustin Y., Khovayko O., Landsman D., Lipman D. J., Madden T. L., Maglott D. R., Ostell J., Miller V., Pruitt K. D., Schuler G. D., Sequeira E., Sherry S. T., Sirotkin K., Souvorov A., Starchenko G., Tatusov R. L., Tatusova T. A., Wagner L., Yaschenko E. Database resources of the National Center for Biotechnology Information. (англ.) // Nucleic Acids Research. — 2007. — January (vol. 35). — P. D5—12. — doi:10.1093/nar/gkl1031. — PMID 17170002. Архивировано 16 ноября 2016 года.
- ↑ Sherry S. T., Ward M., Sirotkin K. dbSNP-database for single nucleotide polymorphisms and other classes of minor genetic variation. (англ.) // Genome Research. — 1999. — August (vol. 9, no. 8). — P. 677—679. — PMID 10447503. Архивировано 23 апреля 2020 года.
- ↑ Полный список организмов можно найти по ссылке: SNP summary. Архивная копия от 16 января 2018 на Wayback Machine
- ↑ Cariaso, Michael. SNPedia: A Wiki for Personal Genomics (англ.) // Bio-IT World. — 2007.
- ↑ Cariaso M., Lennon G. SNPedia: a wiki supporting personal genome annotation, interpretation and analysis. (англ.) // Nucleic Acids Research. — 2012. — January (vol. 40). — P. D1308—1312. — doi:10.1093/nar/gkr798. — PMID 22140107. Архивировано 7 апреля 2016 года.
- ↑ Liu C., Zhang F., Li T., Lu M., Wang L., Yue W., Zhang D. MirSNP, a database of polymorphisms altering miRNA target sites, identifies miRNA-related SNPs in GWAS SNPs and eQTLs. (англ.) // BMC Genomics[англ.]. — 2012. — 23 November (vol. 13). — P. 661—661. — doi:10.1186/1471-2164-13-661. — PMID 23173617. Архивировано 11 октября 2014 года.
- ↑ Drabovich A. P., Krylov S. N. Identification of base pairs in single-nucleotide polymorphisms by MutS protein-mediated capillary electrophoresis. (англ.) // Analytical Chemistry. — 2006. — 15 March (vol. 78, no. 6). — P. 2035—2038. — doi:10.1021/ac0520386. — PMID 16536443. Архивировано 11 июня 2019 года.
- ↑ Griffin T. J., Smith L. M. Genetic identification by mass spectrometric analysis of single-nucleotide polymorphisms: ternary encoding of genotypes. (англ.) // Analytical Chemistry. — 2000. — 15 July (vol. 72, no. 14). — P. 3298—3302. — doi:10.1021/ac991390e. — PMID 10939403. Архивировано 5 июня 2019 года.
- ↑ Altshuler D., Pollara V. J., Cowles C. R., Van Etten W. J., Baldwin J., Linton L., Lander E. S. An SNP map of the human genome generated by reduced representation shotgun sequencing. (англ.) // Nature. — 2000. — 28 September (vol. 407, no. 6803). — P. 513—516. — doi:10.1038/35035083. — PMID 11029002. Архивировано 4 мая 2020 года.
Ссылки
править- Об SNP на страницах NCBI Архивная копия от 2 сентября 2013 на Wayback Machine (недоступная ссылка) Проверено 1 марта 2018.
- NCBI dbSNP database Архивировано 26 января 2006 года.
- HGMD Архивная копия от 28 апреля 2013 на Wayback Machine
- SNPedia Архивная копия от 16 марта 2022 на Wayback Machine
- International HapMap Project
- GWAS Central Архивная копия от 8 марта 2022 на Wayback Machine
- Проект 1000 геномов Архивная копия от 19 октября 2014 на Wayback Machine
- PharmGKB Архивная копия от 13 мая 2013 на Wayback Machine
В другом языковом разделе есть более полная статья Single-nucleotide polymorphism (англ.). |